可变剪接是真核生物在转录后层面调节基因表达的重要机制。其不仅赋予了基因产物的多样性,而且还在多种生理、病理过程中发挥了调节作用。可变剪接取决于剪接因子对pre-mRNA不同剪接位点的选择性识别[1];并且,当前研究表明长非编码RNA(lncRNA)也参与了对pre-mRNA可变剪接的调控[2]。PTBP1是一种识别嘧啶富集区的RNA结合蛋白,最初被认为是一种抑制性的剪接因子;但后续研究表明,PTBP1也可充当可变剪接的促进因子。付向东、薛愿超等学者则利用CLIP-seq等组学手段,发现PTPB1对可变剪接的抑制或促进作用取决于其相对于可变外显子的定位(即“位置效应”理论):当PTBP1的结合位点靠近可变外显子,会抑制可变剪接;若PTBP1的结合位点靠近构成性外显子,则会促进可变剪接[3,4]。尽管如此,介导PTBP1“位置效应”并影响其可变剪接效应的具体机制尚不得而知。
2023年9月19日,beat365李灵团队和空军军医大学赵晓迪/卢瑗瑗/王新团队,以“封面故事(Cover Story)”的形式在Science子刊Science Signaling发表题为The lncRNA MIR99AHG directs alternative splicing of SMARCA1 by PTBP1 to enable invadopodia formation in colorectal cancer cells的研究论文。该研究发现lncRNA MIR99AHG在结直肠癌(CRC)高转移细胞株以及CRC转移灶中异常高表达;解析了MIR99AHG通过PTBP1调控SMARCA1可变剪接、促进CRC细胞侵袭性伪足形成及远端转移的作用和机制;阐明了MIR99AHG以及该可变剪接调节机制在CRC患者预后和治疗中的临床意义。更为重要的是,该研究还揭示了MIR99AHG通过影响PTBP1在SMARCA1 pre-mRNA上的精细定位来调节SMARCA1可变剪接的新机制,为PTBP1调节可变剪接的“位置效应”理论提供了更为细致的分子生物学证据。


研究人员从细胞系、实验动物和CRC临床样本出发,不仅表明MIR99AHG在CRC高转移细胞株和远端转移组织中显著高表达,还证实MIR99AHG可促进CRC的远端转移,并能作为独立的CRC预后分子。课题组随后对MIR99AHG参与CRC转移的生物学效应和效应机制进行了探讨。研究发现,PTBP1是MIR99AHG主要的蛋白质伴侣;MIR99AHG不仅可增加PTBP1的稳定性,还可通过稳定PTBP1增强CRC细胞的转移和侵袭;并且,ISWI染色质重塑蛋白编码基因SMARCA1是MIR99AHG和PTBP1主要的下游调节靶,MIR99AHG和PTBP1可协同调节SMARCA1第13号外显子的可变剪接(skipping-to-inclusion switch),从而增加失活态的SMARCA1的剪接异构体(SMARCA1-L),进而促进侵袭性伪足形成和CRC转移。
在分子机制层面,研究人员利用RAP、RIP-based mapping assay、RNAscope-ISH/IF co-staining、minigene reporter等多研究手段,发现MIR99AHG可以充当PTPB1在SMARCA1 pre-mRNA上的“地址标签”,引导PTBP1结合于靠近第12号构成性外显子的剪接位点,影响了PTBP1在SMARCA1第13号外显子附近的精细定位。这一效应使得PTBP1对可变剪接的调控作用倾向于促进,最终让第13号外显子保留于SMARCA1的mature mRNA。因此,该研究从lncRNA调控的角度为PTBP1调节可变剪接的“位置效应”理论提供了分子生物学解释。此外,研究人员还对调节MIR99AHG表达的上游生物学事件进行了探讨,发现微环境中的肿瘤相关成纤维细胞(CAFs)可分泌TGF-β1,通过TGF受体激活SMAD信号通路,从而上调CRC细胞中的MIR99AHG表达。
总之,该研究不仅揭示了lncRNA MIR99AHG通过剪接因子PTBP1调节SMARCA1可变剪接并参与CRC侵袭性伪足形成及远端转移的新机制(图1),还为PTBP1调节可变剪接的“位置效应”理论提供了分子生物学解释(图2)。领域资深专家吴缅教授(中国科学技术大学)、钟声教授(University of California, San Diego)对本工作进行了点评,认为本研究是对“剪接因子PTBP1位置效应理论”的有益补充。
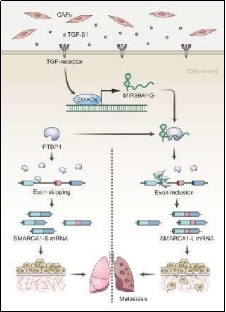
图1
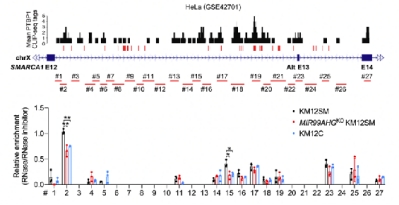
图2
空军军医大学李丹秀、汪鑫博士、beat365手机官方网站助理研究员缪辉博士、空军军医大学刘浩博士为该论文的共同第一作者,空军军医大学第一附属医院赵晓迪、卢瑗瑗副教授、beat365手机官方网站李灵副研究员、空军军医大学第二附属医院王新教授为共同通讯作者。该研究得到了国家自然科学基金优秀青年科学基金项目、国家重点研发计划、四川省科技计划项目、beat365理科原创基金的支持。
1.Campbell ZT, Bhimsaria D, Valley CT, et al. Cooperativity in RNA-protein interactions: global analysis of RNA binding specificity. Cell Rep. 1, 570–581 (2012).
2.Pisignano G, Ladomery M. Epigenetic regulation of alternative splicing: how lncRNAs tailor the message. Noncoding RNA 7, 21 (2021).
3.Xue Y, Zhou Y, Wu T, Zhu T, Ji X, Kwon YS, Zhang C, Yeo G, Black DL, Sun H, Fu XD, Zhang Y. Genome-wide analysis of PTB-RNA interactions reveals a strategy used by the general splicing repressor to modulate exon inclusion or skipping. Mol Cell, 36, 996–1006 (2009).
4.Llorian M, Schwartz S, Clark TA, Hollander D, Tan LY, Spellman R, Gordon A, Schweitzer AC, Grange P, Ast G, Smith CWJ. Position-dependent alternative splicing activity revealed by global profiling of alternative splicing events regulated by PTB. Nat Struct Mol Biol, 17, 1114–1123 (2010).
